Sergi SMIDTAS sergi@sergi5.com
How To Analyse Data from micro-array experiments :
A Simple Tutorial
Source de données
L’analyse commence lorsque les MicroArray ont été scannées. Des images, des logiciels (Genepix, ScanAnalyse..) tirent un tableau de donnée pour chaque spot.
![]()

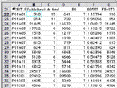
Ce tableau comporte des données relatives au spot :
Diametre
Nombre de pixels
Mediane de l’Intensite 535nm
Mediane de l’Intensité 535nm
Moyenne de l’Intensite 535nm
Moyenne de l’Intensité 535nm
Intenstité Background 635nm
Intenstité Background 535nm
…
Et des données relatives à la qualité du spot moins utilisées :
Flag
Standard déviation du Background
…
Les données telles que le Flag doivent servir à pondérer les données de l’intensité lumineuse des spots par un coefficient compris entre 0 et 1.
0 : spot a ne pas considerer.
1 : très joli spot
Background
Lors du scan d’images, il existe un bruit de fond qui biaise les résultats. Il convient donc de retirer ce bruit de fond. Plusieurs moyens existent pour prendre un tel bruit de fond en considération.
Bruit de fond global
Cette méthode revient à retirer à l’ensemble des spots une valeur unique pour une lame (ou un patch). Le bruit de fond comportant un gradient important sur la lame, cette méthode est limitée.
Bruit de fond local
La plus part des logiciels fournissent à présent pour chaque spot une valeur de bruit de fond local qui consiste à calculer l’intensité moyenne ou médiane d’une zone périphérique à chaque spot.
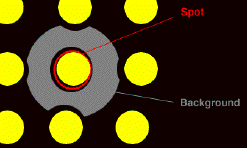
On retire à chaque spot le background pour chaque couleur :
Med535 / BackgroundMed535
Med635 / BackgroundMed635
Médiane, Moyenne, Intensité ?
Les spots comportent plusieurs pixels. Une seule valeur d’intensité sera retenue pour le spot. S’agit-il de la moyenne, de médiane des pixels, ou de l’intensité totale additive des pixels ?
La médiane représente l’intensité du nieme pixel du spot, ou n est la moitie du nombre des pixels du spot classés par intensité. L’avantage est que cette valeur n’est influencée par les pixels d’intensité extrême que par leur nombre, et non leur intensité. Que ce soit les pixels peu nombreux dus à des poussières lumineuses, ou bien les pixels noirs du background qui auraient été pris en compte dans le cercle. L’inconvénient est que cette médiane est influencée par le nombre de pixels du background qui peut être inclus dans le cercle posé par le logiciel de reconnaissance de spots.
La moyenne représente l’intensité moyenne du spot. L’inconvénient est que cette valeur est influencée d’une part, par le background qui pourrait apparaître dans le cercle du spot (lors de spots non circulaires et lors de cercles de taille fixes) et d’autre part, par les poussières.dont l’intensité est très importante.
L’intensité n’est pas fournie en général par les logiciels d’analyse. L’intensité=(Nombre de pixel )*(Valeur Moyenne). Elle représente la quantité de lumière émise par le spot, et a l’avantage de mieux correspondre à la quantité de luminophores présente dans le spot.
L’autre avantage est qu’elle permet d’utiliser de grands cercles pour le repérage des spots, puisque si une partie du background est dans le cercle, ce dernier n’aura aucune influence. Le repérage des spots se fait alors plus facilement.
L’inconvénient est que la quantité de lumière due aux poussières est également prise en compte.

Intens. Excellent Excellent Excellent Excellent Bad Excellent
Med Good Poor Poor Poor Good Good
Mean Good Bad Bad Poor Poor Poor
Nous verrons plus loin comment comparer ces 3 méthodes de manière plus quantitative et systématique.

Black Hole
Les trous noirs introduisent des intensités négatives lorsque l’on retire le bruit de fond. Une solution consiste à ne pas tenir compte de ces spots dans l’analyse. :-(
Représentation de données
Le logarithme est intéressant pour travailler sur les intensités obtenues, et rendre visibles les spots. De faible intensité.
Le logarithme introduit, il faudra faire attention dans la suite qu’une division avant log équivaut à une soustraction.
log(Cy3/Cy5)=log(Cy3)-log(Cy5)
log(Cy3*Cy5)=log(Cy3)+log(Cy5)
Ainsi, si le background n’a pas encore été retiré, c’est en soustrayant sa valeur et non par une division que l’on y arrivera.
log(Rouge)=log(Cy5)-log(Background)=log(Cy5/Background)
log
![]()
![]()
Que le logarithme soit népérien ( ln(2,718)=1 ), décimal ( log(10)=1 ) ou en base 2 (log2(2)=1 ) les résultats seront inchangés à un rapport prêt. Leur représentation sera identique.Il faut tout de même savoir quel logarithme a été utilisé pour tirer des conclusions quantitatives d’expérience.
Ainsi,un ratio logarithmique de avec un log correspont à un rapport Cy/Ref de
1 décimal 10
0,3 décimal 2
1 néperien 2,718
0,4 népérien 1,5
0,7 népérien 2
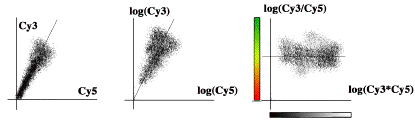
Ratio et Intensité sont bien plus significatifs qu’intensité rouge contre intensité verte et il convient par conséquent plus agréable de les porter sur les axes.
Ratio = log(Cy3/Cy5) ou log(Cy5/Cy3)
Intensity = log(Cy3*Cy5)
La lecture des graphes est alors immédiate et naturelle.
![]()
![]()
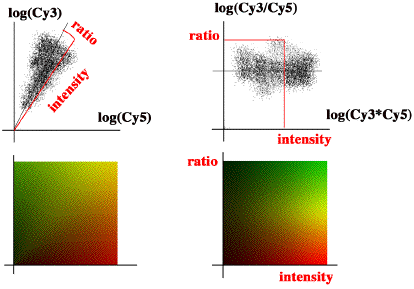
Le ratio d’un gène sur-exprimé sera positif ( log(Cy/Ref)>0 )
Le ratio d’un gène sous-exprimé sera négatif ( log(Cy/Ref)>0 )
Normalisation Intra-lame
Lorsque les lames sont scannées, l’intensité dans une des couleurs, n’est pas toujours comparable aux intensités dans l’autre couleur. Pour y remédier le meilleur moyen est d’ajuster la puissance des lasers du scanner pour obtenir la meilleure image sans toute fois saturer aux point les plus lumineux.
![]()
![]()
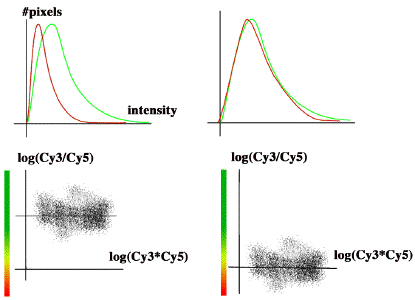
Apres avoir scanné l’image, il est encore possible de normaliser l’intensité d’un fluorophore contre un autre.
Calculer la moyenne des ratios de spots, et retrancher cette valeur à tous les spots.
Ratio=log(Cy/Ref)-Mean[log(Cy/Ref),…]
Cette normalisation peut être effectuée en considérant la moyenne
sur tous les spots
les 80 % de spots médians
certains spots étalons
La valeur moyenne du ratio sera alors approximativement de 0.
De même la normalisation de l’intensité peut être effectuée en considérant des spots étalons.
Normalisation Inter-lame
Pour comparer des lames différentes, ou quelques spots sont censés s’exprimer différemment, il peut être judicieux d’avoir la même déviation standard pour tous les nuages.
Comme pour la normalisation intra-lame, cette déviation standard peut être calculée
sur l’ensemble des spots
les 90% des spots médians
une sélection de spots
On s’intéresse généralement plus souvent au ratio qu’à l’intensité mais ces normalisations devraient aussi être effectuées sur l’intensité.
![]()
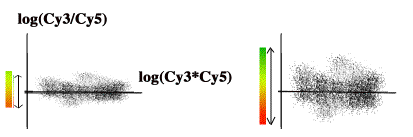
Cette normalisation ne peut être souhaitable que pour des lames comparables. En effet la dynamique de toutes les lames n’est pas forcément identique.
Pour normaliser ainsi les lames, il convient de diviser les ratios par la déviation standard calculée.
Ratio_normalisé = ratio / std[ratios]
Ce qui revient à peu près à
log(Ratio_normalisé) = log(ratio) - std[log(ratios)]
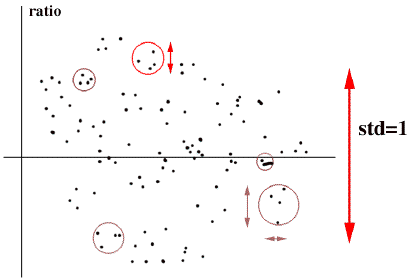
Le résultat obtenu est un nuage de points centrés en 0 par la normalisation intra-lame et de déviation standard 1 par la normalisation inter-lame.
Utilisation de répliquas et reproductibilité
Un échantillon d’adn peut être spoté en plusieurs exemplaires sur une lame. Ces spots sont reconnus séparément par les logiciels de reconnaissance de spots. Il faut à un moment regrouper ces spots pour en tirer la signification particulière qu’ils procurent.
Disposer de plusieurs mesures pour les résultats d’hybridation d’un même échantillon d’adn améliore la précision des résultats. La moyenne des ratios mesurés d’un répliqua donne une bonne estimation du ratio pour un échantillon d’adn hybridé.
Voici un ordre de grandeur pour une lame de déviation standard normalisée à 1 :
Erreur(norm.) Nbre Spots à plus de 1,5 fois la valeur considérée
1 spot par répliqua 0,92 80%
2 spots par répliqua 0,74 89%
3 spots par répliqua 0,62 92%
4 spots par répliqua 0,57 94%
Cela signifie que, si l’on utilise 3 spots par répliqua, 8 % des ratios obtenus seront en réalité à l’extérieur d’une fourchette de 1,5 fois autour de la valeur mesurée.
On est 2 fois plus précis en passant de 1 à 2 spots par répliqua, et 4 fois plus précis avec 4 spots par répliqua.
Quand on change de lame, les résultats se dégradent.
Estimation de l’erreur (en log népérien)
1 spot sur chaque lame par répliqua 2,56
2 spots sur chaque lame par répliqua 2,10
4 spots sur chaque lame par répliqua 0,76
L’interet d’utiliser 4 spots par répliqua et par lame est tout à fait notable cette fois.
Localisations des spots sur la lame
Il convient de séparer spatialement le plus possible les spots d’un répliqua sur la lame pour décorreler au maximum les perturbations qui interviennent.
Comparaisons de lames,
Spécificité.
Comparaison de protocoles et qualité
On considère ici que l’on a 2 séries de résultats à comparer. Ces 2 séries différent par une normalisation différente, un protocole différent… mais l’ARN hybridé utilisé est identique dans les 2 cas.
Si l’ARN n’est pas identique, cette méthode peut égalament être utilisée avec précautions car elle fait normalement intervenir une normalisation inter-lame.
La série de résultat sera d’autant meilleur que que les spots de répliquas seront regroupés dans le nuage, ce qui sera un bon indice de la reproductibilité et de la précision des résultats.
On calcule d’une part la déviation Standard des spots au sein des répliqua, et d’autre part la déviation standard de l’ensemble du nuage. Leur différence est caractéristique de la qualité des résultats.
(Std nuage) - (Std répliqua), grand => meilleurs protocole, meilleur reproductibilité.
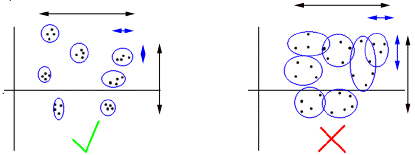
Pour calculer la déviation standard moyenne des spots au sein des répliquas, on calcule pour chaque répliqua la déviation standard. Puis on prend la moyenne des valeurs ainsi obtenues.
Pour calculer la déviation standard du nuage, on prend la déviations standard de toutes les valeurs.
Exemple : Soit à répondre à la question:
« Est ce mieux de considérer la médiane ou l’intensité totale pour étudier l’intensité des spots ? »
On a ici une seule lame, mais avec 2 protocoles différents d’analyse de donnée. Ce qui nous donne nos deux séries de valeurs à comparer.
1. En considérant la Mediane des pixels des spots
2. En considérant l’intensité totale (Rayon*Moyenne)
Afin de déterminer le meilleur protocole des deux, on calcule comme illustrée ci-dessus, la moyenne des déviations standards de l’intensité des spots pour le nuage et par répliqua.
Pour le 1er protocole : (les valeurs sont en logarithme népérien)
Standard déviation dans les répliquas : 1,24
Standard déviation du nuage : 2,24
Soit une différence: 1 (2,24 - 1,24 = 1)
Pour le 2eme protocole :
Standard déviation dans les répliquas : 1,38
Standard déviation du nuage : 2,45
Soit une différence: 1,07 (2,45 - 1,38 = 1,07)
Ce qui permet de conclure comme attendu (cf explication données plus haut) que l’intensité globale est mieux adaptée que la médiane pour l’étude de l’intensité des spots.
Comparaison de gènes
Il existe des méthodes de clustering pour comparer plusieurs lames, non abordées ici. Ces méthodes regroupent les gènes suivant une ressemblance de leurs vecteurs, profil d’expression, à une ou plusieurs dimensions.
Cependant dans le cas de 2 lames seulement, il peut être plus intéressant de comparer directement les lames en regardant leur nuage de répliqua. (les moyennes des spots par répliquas des lames normalisées ayant déjà été prises en compte).

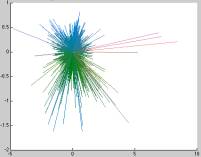
En reliant sur le nuage (Ratio en fonction de intensité) par des traits les valeurs d’une lame à l’autre pour chaque répliqua, on obtient une représentation qui permet de donner une idée de ce qui se passe, sans toute fois être exploitable. Cette représentation a néanmoins l’avantage de mettre l’accent sur la représentatitivité des résultats, et sur l’importance du bruit expérimental sur les valeurs mesurées. (graphe ci dessus en suivant les spots et non les répliquas, d’ou une image peu lisible)
En relativisant la variation d’expression des gènes par rapport à une lame, (et sous forme de nuage de points ) les gènes variant le plus sont tout de suite repérable,puisqu’ils sont les représentés par des points, les plus loints du centre. Cette représentation a l’avantage de ne pas être perturbée visuellement par le bruit de fond de variation dynamique de l’expression de gènes.( graphe ci-dessous avec des traits au lieu de l’avoir en nuage de points)